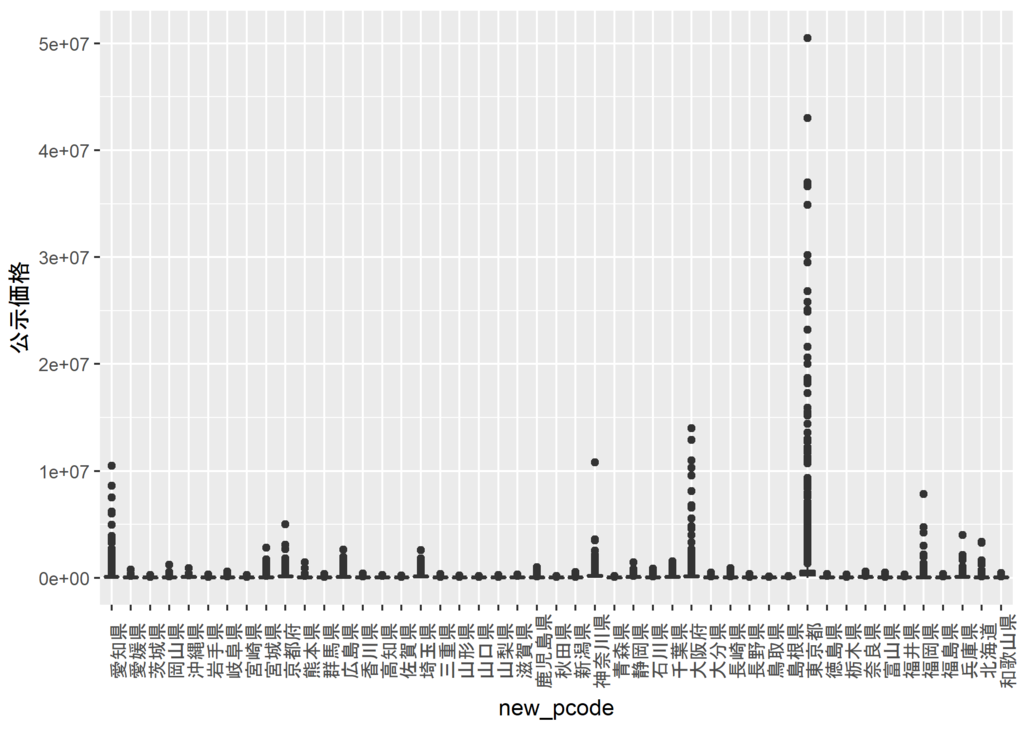
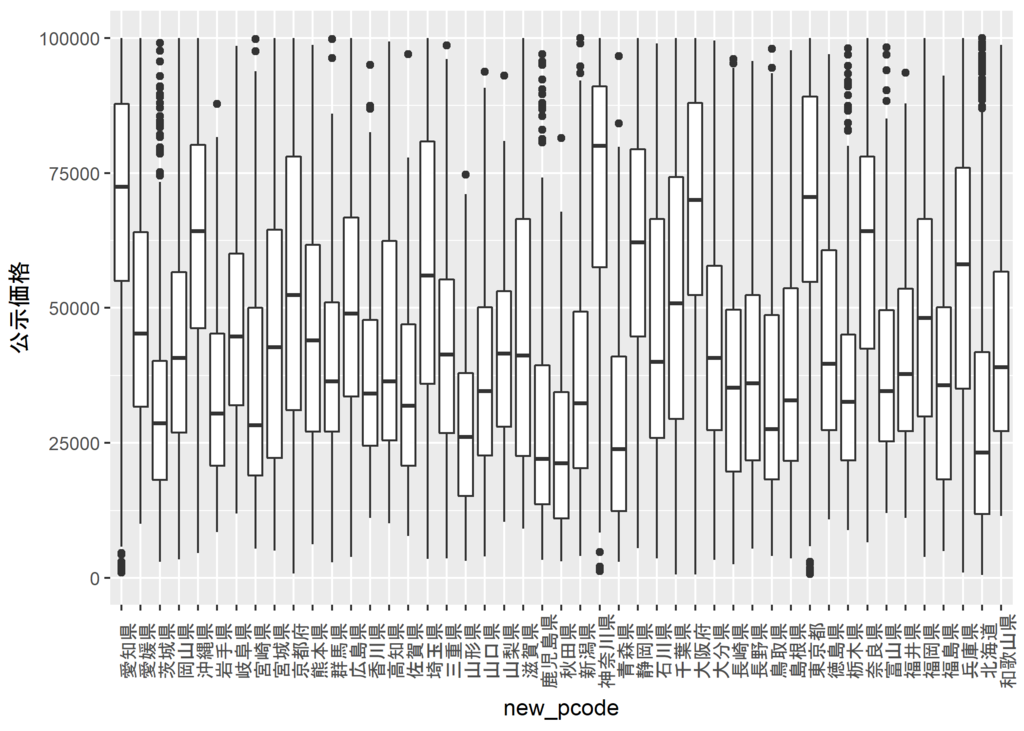
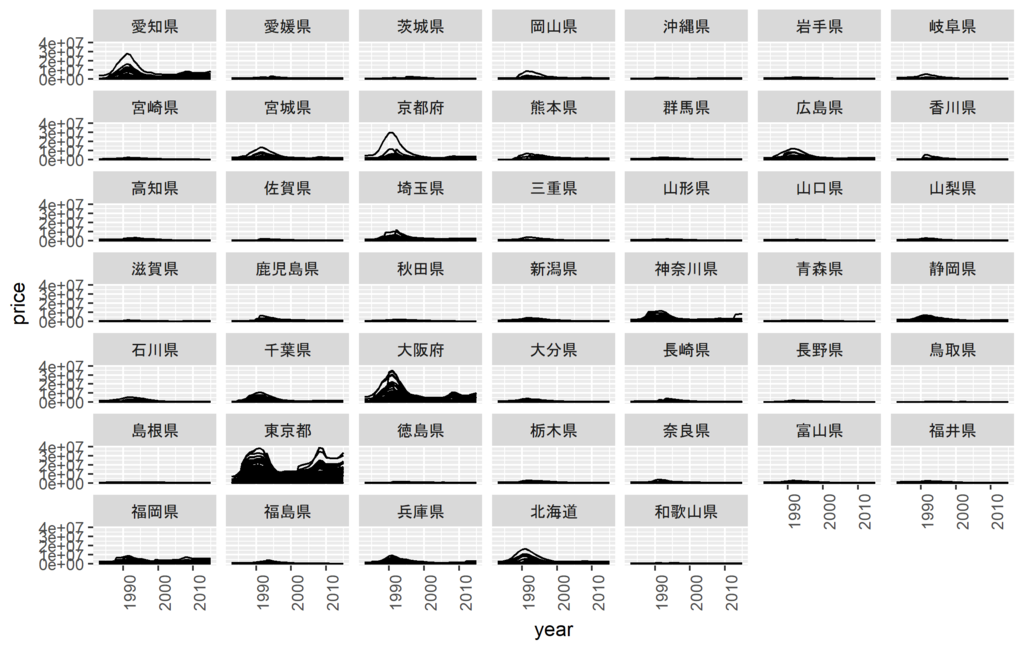
data.frameからsfクラスのデータを作成する方法
結論から言うとst_as_sf(data.frame, coords=c(x, y), crs=4612)というように行えばよい
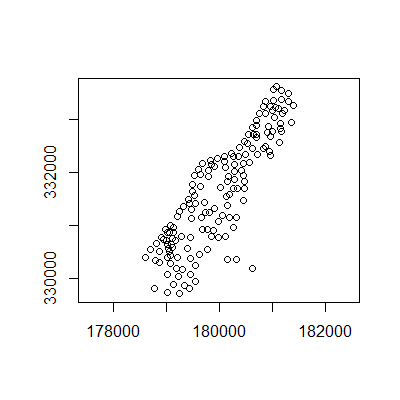
library(sp) library(sf) data(meuse)
head(meuse) ## x y cadmium copper lead zinc elev dist om ffreq soil lime landuse ## 1 181072 333611 11.7 85 299 1022 7.909 0.00135803 13.6 1 1 1 Ah ## 2 181025 333558 8.6 81 277 1141 6.983 0.01222430 14.0 1 1 1 Ah ## 3 181165 333537 6.5 68 199 640 7.800 0.10302900 13.0 1 1 1 Ah ## 4 181298 333484 2.6 81 116 257 7.655 0.19009400 8.0 1 2 0 Ga ## 5 181307 333330 2.8 48 117 269 7.480 0.27709000 8.7 1 2 0 Ah ## 6 181390 333260 3.0 61 137 281 7.791 0.36406700 7.8 1 2 0 Ga ## dist.m ## 1 50 ## 2 30 ## 3 150 ## 4 270 ## 5 380 ## 6 470
meuse_sf <- st_as_sf(meuse, coords = c("x", "y"), crs = 28992)
class(meuse_sf) ## [1] "sf" "data.frame"
meuse_sf %>% st_geometry() %>% plot(axes=T)
sfクラスのデータ抽出
ポリゴン内のポイントまたはポイントがあるポリゴンデータを抽出したい場合がある.
特に難しいことはないが,メモとして残しておく.
library(sf) set.seed(1) nc <- system.file("shape/nc.shp", package="sf") %>% st_read nc1 <- nc[1:50, ] nc_points <- st_sample(nc, size = 10) # plot(nc) plot(st_geometry(nc), border = "grey40") # plot(nc1) plot(st_geometry(nc1),border="grey40", col = "grey90", add = T) # plot(nc_points) plot(nc_points, add =T, pch = 16) # plot(nc1_filtered) nc1[nc_points, ] %>% plot(add=T, lwd = 2, border = "blue") # plot(nc_points_filtered) nc_points[nc1] %>% plot(add = T, col = "violet", cex = 3, lwd = 2)

公示価格を可視化してみた
popupTable()の利用に関して
mapview::popupTable()を利用すると、簡単にポップアップを作成でき便利であるが、 windows環境だと文字コードの問題があり?うまく表示できないことが多い。
その際には以下のようにすることで解決することができる。
m_base <- leaflet(options = leafletOptions(crs = leafletCRS(code = "EPSG:4612"))) %>% addTiles() m_base %>% addPolygons(data = N03, popup = mapview::popupTable(N03) %>% lapply(enc2utf8))
J-STAGEのWebAPIを利用してみる
J-STAGE WebAPIを利用してみる
r Sys.Date()
J-stageからWebAPIを利用して論文タイトル等の一覧を取得する
J-STAGEは普段使うにはアクセスに時間がかかり使いにくいと思っていたが、 J-STAGE WebAPI が利用できるようなので試してみる。
建築学会の論文集を取得し、各界の権威?を調べてみる。
library(rvest) library(tidyverse)
巻号取得の際に指定できるパラメータは以下のようになっている。
論文検索結果取得の際とパラメータ指定が異なるので、注意する。
また、論文検索の場合は1,000件が上限のようなので、超える場合は工夫して取得する必要がある。
| No. | parameter | 必須・任意 | 内容 | 備考 |
|---|---|---|---|---|
| 1 | service | 必須 | 利用する機能を指定 | 巻号一覧取得は2、論文検索結果取得は3を指定 |
| 2 | system | 任意 | 検索対象のシステムを指定 | 廃止 |
| 3 | pubyearfrom | 任意 | 発行年を指定(from) | 西暦4桁 |
| 4 | pubyearto | 任意 | 発行年を指定(to) | 西暦4桁 |
| 5 | material | 任意 | 資料名の検索語句を指定 | 完全一致検索 |
| 6 | issn | 任意 | onlineISSN またはPrintISSNを指定 | 完全一致検索 xxxx-xxxx形式 |
| 7 | cdjournal | 任意 | 資料コードを指定 | J-STAGEで付与される資料を識別するコード |
| 8 | volorder | 任意 | 巻の並び順を指定 | 1:昇順, 2:降順, 未指定時は1 |
建築学会の論文集一覧を取得際の
ONLINE ISSNは以下のようになっている。
構造系:1881-8153
環境系:1881-817X
計画系:1881-8161
# データを取得するパラメータの設定 url_base1 <- "http://api.jstage.jst.go.jp/searchapi/do?service=3&pubyearfrom=2010&" url_base2 <- "http://api.jstage.jst.go.jp/searchapi/do?service=3&pubyearfrom=2010&start=1001&count=1000&" url_base3 <- "http://api.jstage.jst.go.jp/searchapi/do?service=3&pubyearfrom=2010&start=2001&count=1000&" url_structure <- "issn=1881-8153" url_environment <- "issn=1881-817X" url_plan <- "issn=1881-8161" # データの取得 res_s1 <- read_xml(paste0(url_base1, url_structure)) res_e1 <- read_xml(paste0(url_base1, url_environment)) res_p1 <- read_xml(paste0(url_base1, url_plan)) res_s2 <- read_xml(paste0(url_base2, url_structure)) res_e2 <- read_xml(paste0(url_base2, url_environment)) res_p2 <- read_xml(paste0(url_base2, url_plan)) res_p3 <- read_xml(paste0(url_base3, url_plan))
# データの整形 get_data <- function(res = res){ #res <- read_xml(paste0(url_base, url_structure)) title <- res %>% xml_nodes(xpath = "d1:entry/d1:article_title/d1:ja") %>% xml_text() author_ja <- res %>% xml_nodes(xpath = "d1:entry/d1:author/d1:ja") %>% sapply(., function(x) {x %>% xml_contents %>% xml_text}) %>% plyr::ldply(rbind) %>% `colnames<-`(paste("name_ja", colnames(.), sep = "")) author_en <- res %>% xml_nodes(xpath = "d1:entry/d1:author/d1:en") %>% sapply(., function(x) {x %>% xml_contents %>% xml_text}) %>% plyr::ldply(rbind) %>% `colnames<-`(paste("name_en", colnames(.), sep = "")) pubyear <- res %>% xml_nodes(xpath = "d1:entry/d1:pubyear") %>% xml_text() volume <- res %>% xml_nodes(xpath = "d1:entry/prism:volume") %>% xml_text() number <- res %>% xml_nodes(xpath = "d1:entry/prism:number") %>% xml_text() stPage <- res %>% xml_nodes(xpath = "d1:entry/prism:startingPage") %>% xml_text() d <- data.frame(title, author_ja, author_en, pubyear, volume, number, stPage) # 変なデータを除去する(日本語英語いずれの著者名に対してデータない場合は除去する d <- d %>% filter(!is.na(name_ja1) | !is.na(name_en1)) return(d) } d_s1 <- get_data(res_s1) d_e1 <- get_data(res_e1) d_p1 <- get_data(res_p1) d_s2 <- get_data(res_s2) d_p2 <- get_data(res_p2) d_p3 <- get_data(res_p3) d_s <- bind_rows(d_s1, d_s2) d_e <- d_e1 d_p <- bind_rows(d_p1, d_p2, d_p3) d_s <- d_s %>% mutate(field = "structure") d_e <- d_e %>% mutate(field = "environment") d_p <- d_p %>% mutate(field = "plan") d <- bind_rows(d_s, d_e, d_p)
やってみた
論文の発行数を確認する
少子高齢化?学術界の減退?により近年論文数は減ってきている。
d %>% group_by(field, pubyear) %>% tally() %>% ggplot(aes(x = pubyear, y = n, color = field, group = field)) + geom_line() + geom_point()
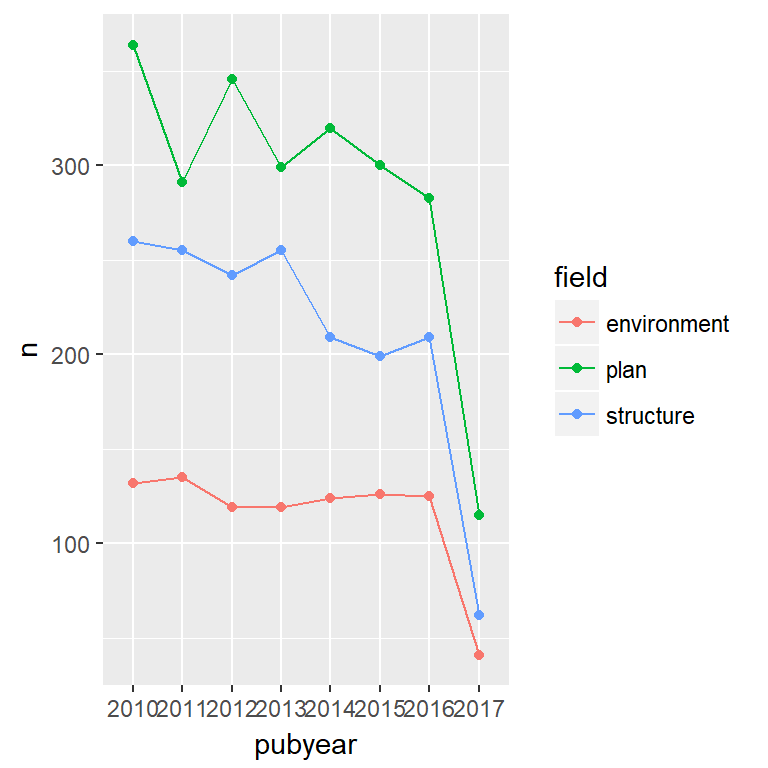
各分野で投稿数が多い人順に並び変える
単純に論文に多くかかわっている人を確認してみる。
d %>% select(starts_with("name_en"), field) %>% gather(num_author, value = name, -field) %>% filter(is.na(name) == F) %>% group_by(name, field) %>% summarise(n_submit = n()) %>% ungroup %>% group_by(field) %>% top_n(n = 5) %>% arrange(field, desc(n_submit))
Source: local data frame [15 x 3]
Groups: field [3]
name field n_submit
<chr> <chr> <int>
1 Shinsuke KATO environment 43
2 Shin-ichi TANABE environment 28
3 Hirofumi HAYAMA environment 26
4 Toshiharu IKAGA environment 25
5 Ryozo OOKA environment 24
6 Mitsuo TAKADA plan 39
7 Takeshi NAKAGAWA plan 28
8 Teruyuki MONNAI plan 27
9 Haruhiko GOTO plan 25
10 Keisuke KITAGAWA plan 22
11 Kazuhiko KASAI structure 44
12 Toru TAKEUCHI structure 41
13 Hitoshi KUWAMURA structure 38
14 Satoshi YAMADA structure 36
15 Shoichi KISHIKI structure 32
d %>% select(starts_with("name_ja"), field) %>% gather(num_author, value = name, -field) %>% filter(is.na(name) == F) %>% group_by(name, field) %>% summarise(n_submit = n()) %>% ungroup %>% group_by(field) %>% top_n(n = 10) %>% arrange(field, desc(n_submit))
Source: local data frame [32 x 3]
Groups: field [3]
name field n_submit
<chr> <chr> <int>
1 加藤 信介 environment 44
2 羽山 広文 environment 29
3 田辺 新一 environment 29
4 伊香賀 俊治 environment 25
5 大岡 龍三 environment 24
6 西名 大作 environment 22
7 村上 周三 environment 21
8 吉野 博 environment 20
9 村川 三郎 environment 20
10 伊藤 一秀 environment 18
# ... with 22 more rows
年度も考慮してみる
最近は出していないのかや最近出すようになったのかなど、 いつ頃論文を多く出したのか確認できる。
d %>% select(starts_with("name_ja"), field, pubyear) %>% gather(key = num_author, value = name, -field, -pubyear) %>% filter(is.na(name) == F) %>% group_by(name, field, pubyear) %>% tally() %>% ungroup %>% group_by(name, field) %>% mutate(totalN = sum(n)) %>% group_by(field) %>% spread(key = pubyear, value = n) %>% top_n(n = 10, wt = totalN) %>% arrange(field, desc(totalN))
Source: local data frame [32 x 11]
Groups: field [3]
name field totalN `2010` `2011` `2012` `2013` `2014`
<chr> <chr> <int> <int> <int> <int> <int> <int>
1 加藤 信介 environment 44 11 9 6 6 4
2 羽山 広文 environment 29 5 1 3 2 1
3 田辺 新一 environment 29 4 3 1 5 5
4 伊香賀 俊治 environment 25 3 5 4 3 2
5 大岡 龍三 environment 24 5 4 3 1 3
6 西名 大作 environment 22 NA 1 1 6 8
7 村上 周三 environment 21 3 6 4 4 2
8 吉野 博 environment 20 2 3 4 4 3
9 村川 三郎 environment 20 NA 1 5 7 6
10 伊藤 一秀 environment 18 4 4 NA 2 1
# ... with 22 more rows, and 3 more variables: `2015` <int>, `2016` <int>,
# `2017` <int>
ファーストオーサーで投稿数が多い人を確認する
ここで出てきた人は論文を書く能力が高い、 もしくはまとめる能力が高い人といえるかもしれない。
d %>% select(starts_with("name_en"), field) %>% gather(num_author, value = name, -field) %>% filter(num_author == "name_en1") %>% filter(is.na(name) == F) %>% group_by(name, field) %>% summarise(n_submit = n()) %>% ungroup %>% group_by(field) %>% top_n(n = 10) %>% arrange(field, desc(n_submit))
Source: local data frame [32 x 3]
Groups: field [3]
name field n_submit
<chr> <chr> <int>
1 Go IWASHITA environment 9
2 Toshimasa KAWANISHI environment 9
3 Yasushi KONDO environment 9
4 Hideki KIKUMOTO environment 7
5 Hideki TAKEBAYASHI environment 7
6 Jun CUI environment 6
7 Kensuke KOBAYASHI environment 6
8 Koki KIKUTA environment 6
9 Takaho ITOIGAWA environment 6
10 Tomoaki NISHINO environment 6
# ... with 22 more rows
d %>% select(starts_with("name_ja"), field) %>% gather(num_author, value = name, -field) %>% filter(num_author == "name_ja1") %>% filter(is.na(name) == F) %>% group_by(name, field) %>% summarise(n_submit = n()) %>% ungroup %>% group_by(field) %>% top_n(n = 10) %>% arrange(field, desc(n_submit))
Source: local data frame [35 x 3]
Groups: field [3]
name field n_submit
<chr> <chr> <int>
1 岩下 剛 environment 9
2 近藤 靖史 environment 9
3 川西 利昌 environment 9
4 菊本 英紀 environment 7
5 竹林 英樹 environment 7
6 菊田 弘輝 environment 6
7 糸井川 高穂 environment 6
8 小林 謙介 environment 6
9 西野 智研 environment 6
10 崔 軍 environment 6
# ... with 25 more rows
ファーストオーサー以外で著者数を調べる
ここに出てきた人は論文を書かせてくれる(もしくは書かされる)、 人脈が広く、様々なことに挑戦しているてる人といえるかもしれまい。
d %>% select(starts_with("name_ja"), field) %>% gather(num_author, value = name, -field) %>% filter(num_author != "name_ja1") %>% filter(is.na(name) == F) %>% group_by(name, field) %>% summarise(n_submit = n()) %>% ungroup %>% group_by(field) %>% top_n(n = 10) %>% arrange(field, desc(n_submit))
Source: local data frame [30 x 3]
Groups: field [3]
name field n_submit
<chr> <chr> <int>
1 加藤 信介 environment 44
2 羽山 広文 environment 29
3 田辺 新一 environment 29
4 伊香賀 俊治 environment 24
5 大岡 龍三 environment 24
6 西名 大作 environment 22
7 村上 周三 environment 21
8 村川 三郎 environment 20
9 浅野 良晴 environment 18
10 吉野 博 environment 17
# ... with 20 more rows
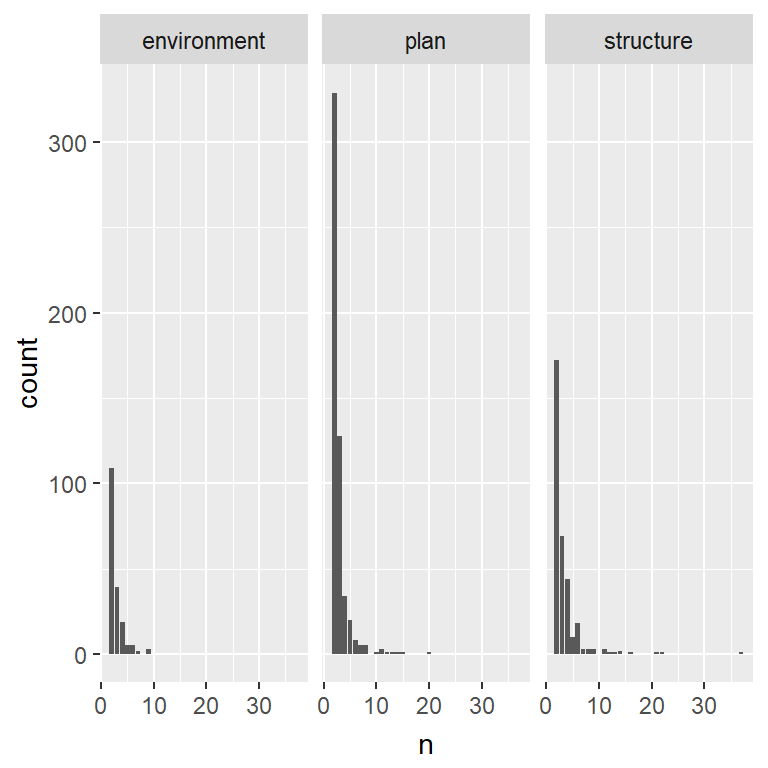
論文投稿数を確認する
各分野ごとに掲載数を確認してみる。
d %>% select(starts_with("name_ja"), field) %>% gather(num_author, value = name, -field) %>% filter(num_author == "name_ja1") %>% filter(is.na(name) == F) %>% group_by(name, field) %>% summarise(n = n()) %>% ggplot(aes(x = n))+ geom_bar() + facet_wrap(~field)

一本のみの掲載の人の割合を確認してみる。
d %>% select(starts_with("name_ja"), field) %>% gather(num_author, value = name, -field) %>% filter(num_author == "name_ja1") %>% filter(is.na(name) == F) %>% group_by(name, field) %>% summarise(n = n()) %>% mutate(one = {n == 1}) %>% xtabs(~one+field, data = .)
field one environment plan structure FALSE 182 538 333 TRUE 414 800 552
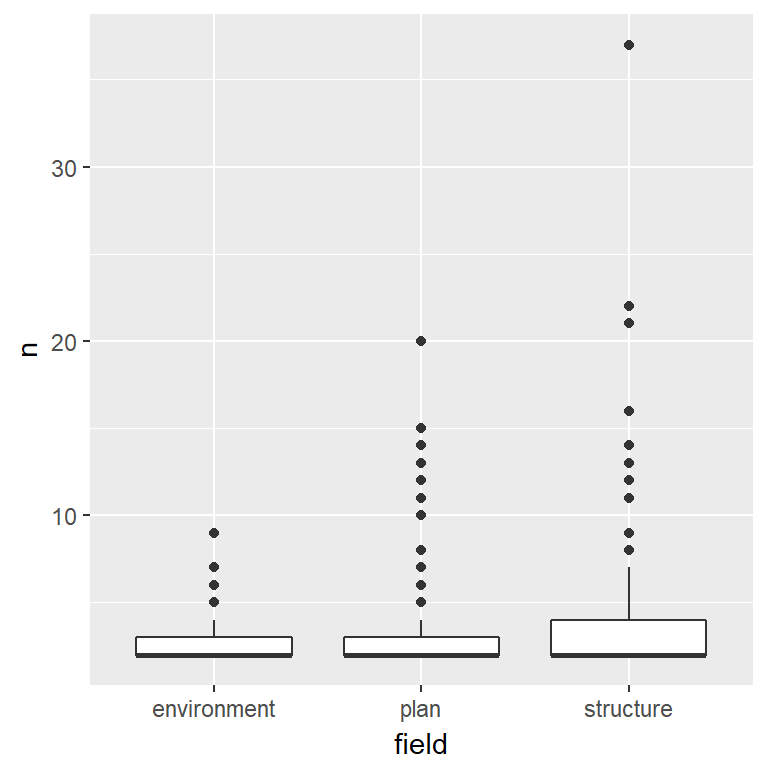
投稿が一本のみの人を除外した場合の投稿数の分布
d %>% select(starts_with("name_ja"), field) %>% gather(num_author, value = name, -field) %>% filter(num_author == "name_ja1") %>% filter(is.na(name) == F) %>% group_by(name, field) %>% summarise(n = n()) %>% ungroup %>% filter(n != 1) %>% ggplot(aes(x = n))+ geom_bar() + facet_wrap(~field)
d %>% select(starts_with("name_ja"), field) %>% gather(num_author, value = name, -field) %>% filter(num_author == "name_ja1") %>% filter(is.na(name) == F) %>% group_by(name, field) %>% summarise(n = n()) %>% ungroup %>% filter(n != 1) %>% ggplot(aes(x = field, y = n))+ geom_boxplot()
RでのGIS演算時には座標参照系の確認が大切
RでGIS演算(例えば面積を算出)を行う場合には座標参照系を確認しておく必要がある。
adm_area <- sf::st_read("N03/2016/N03-16_17_160101.shp", crs = 4612, options = "ENCODING=UTF-8")
adm_area %>% as(., "Spatial") %>% rgeos::gArea(byid = T) %>% .[1]
出力結果: 3.137912e-06
adm_area %>% as(., "Spatial") %>% `proj4string<-`("+init=epsg:4612") %>% sp::spTransform("+init=epsg:2449") %>% rgeos::gArea(byid = T) %>% .[1]
出力結果:31140.31
adm_area %>% mutate(area = sf::st_area(.)) %>% .[1,]
出力結果: 31144.56 m^2
sf::st_area()はCRSを変換しなくても算出してくれるようだが、 安全のため変換しておく癖をつけておいたほうがよいだろう。